Projekte
Einzelmolekül Fluoreszensresonanzenergietransfer (smFRET) Experimente nutzen die Abstandsabhängigkeit der messbaren Transfereffizienz zwischen zwei Farbstoffen aus, um Abstände auf der nm Skala zu bestimmen. Zusätzlich hängt die Transfereffizienz jedoch auch von der relativen Orientierung der Farbstoffe ab, welche in Experimenten nicht direkt bestimmt werden kann. In Moleküldynamik-Simulationen ist die relative Farbstofforientierung hingegen bestimmbar. Daher ermöglicht die Kombination von Simulationen mit Experimenten eine genauere Abstandsbestimmung als die ausschließlichen Bestimmung aus Experimenten.

FRET Näherung
Einzelmolekül Fluoreszensresonanzenergietransfer (smFRET) Experimente messen den strahlungsfreien Energietransfer von einem Donor- zu einem Akzeptorfarbstoff. Die Transfereffizienz hängt vom Abstand der beiden Farbstoffe ab und kann durch Messung der relativen Fluoreszensintensitäten von Donor und Akzeptor bestimmt werden. Wird die FRET Effizienz von einem einzelnen mit Farbstoffen markierten Molekül gemessen, so können Abstände im Molekül oder zwischen molekularen Untereinheiten bestimmt werden. Das optische Auslesen der Abstandsinformation ermöglicht die Anwendung der Technik bei Raumtemperatur und -druck sowie Messungen in lebenden Zellen. Neueste Entwicklungen bei zeitaufgelösten (time-resolved: tr) Experimenten ermöglichen die Untersuchung der Einzelmoleküldynamik. Durch diese Vorteile wurde smFRET zu einem vielseitigen Werkzeug in der Biophysik für Messungen im Nanometer Bereich um biologische Strukturen und ihre Funktion zu untersuchen. Ligand-Rezeptor Wechselwirkungen, Konformationsänderungen, Proteinsynthese, Faltungskinetik und Gen-Translokation sind nur einige der Vielzahl von Problemen, die mit tr-smFRET studiert werden können.
Analog zum Rundfunk, bei dem die Empfangsstärke auch von der Antennenausrichtung abhängt, ist auch die Transfereffizienz nicht nur abhängig vom Abstand sonder auch von der relativen Farbstofforientierung. Daher ist die Bestimmung von Abständen aus smFRET Experimenten nicht trivial und benötigt zusätzliche Näherungen und Modellannahmen. Typische Näherungen sind eine isotrope und unkorrelierte Orientierungsverteilung der Übergangsdipolmomente, sowie die Annahme, daß die Kopplungsstärke der Übergangselektronendichten hinreichend genau durch Dipol-Dipol Kopplung beschrieben ist. Mit diesen und weiteren Annahmen bestimmte Theodor Förster die Transfereffizienz [1] zu \[E(R) = \frac{1}{1+\left(\frac{R}{R_0}\right)^6} \qquad (1),\] wobei \(R\) der Abstand der Farbstoffe und \(R_0\) der Abstand mit 50 % Anregungstransfer ist. Die Orientierungsabhängigkeit der Dipol-Dipol Kopplung wird beschrieben durch \[\kappa=\cos\theta_{DA}-3\cdot\cos\theta_D\cdot\cos\theta_A \qquad (2),\] mit \(\theta_{DA}\) als Winkel zwischen den Übergangsdipolmomenten der Farbstoffe. \(\theta_D\) und \(\theta_A\) sind die jeweiligen Winkel zwischen Donor- und Akzeptorübergangsdipolmoment und dem Verbindungsvektor \(R\) der Farbstoffe. Der Förster Radius \(R_0^6\) ist \(\propto \kappa^2\) und \(\langle\kappa^2\rangle=\frac{2}{3}\) für isotrope Orientierungsverteilungen.
An Biomolekülen angebrachte Farbstoffe sind durch das Biomolekül räumlich eingeschränkt und wechselwirken mit dessen Oberfläche. In der Tat wurden große Abweichungen von isotropen Orientierungsverteilungen gefunden, begleitet von einer Abweichung des Orientierungsfaktors \(\langle\kappa^2\rangle\) von \(\frac{2}{3}\) [2]. Die genaue Bestimmung von Abständen mittels FRET wird begrenzt durch die Ungewissheit über die Farbstofforientierung. Im Gegensatz zu Experimenten ist in Moleküldynamik (MD) Simulationen die Farbstofforientierung zu jedem Zeitpunkt zugänglich.
Farbstoffdynamik und FRET Effizienzverteilungen aus MD Simulationen
Um die Genauigkeit der Abstansrekonstruktion zu verbessern entwickelten wir eine Methode zur Kombination von Farbstofforientierungsdynamik und -statistik aus MD Simulationen mit in Experimenten gemessenen FRET Effizienzverteilungen [3]. Dabei wurde Poly-Prolin mit angebrachten Farbstoffen an den Enden als Testsystem untersucht.. Poly-Proline bilden in Wasser eine stabile Typ II Helix und wurden zudem in vielen FRET Experimenten in der Vergangenheit untersucht [4,5]. Um die kinetischen Pfade von smFRET Experimenten abzubilden entwickelten wir eine Markov Ketten Monte Carlo Methode, welche die aus MD Trajektorien bestimmten zeitabhängigen FRET Raten verwendet.
Beispielpfade der Markov Chain Monte Carlo Methode. Im ersten Fall sendet der angeregte Donor nach einiger Zeit ein Photon aus. Im zweiten Fall wird die Anregung mittels Resonanzenergietransfer zum Akzeptor übertragen, welcher diese dann ebenfalls nach einiger Zeit als Photon aussendet.
Photonen Bursts, welche auch im Experiment gemessen werden, wurden unter Verwendung der entwickelten Markov Chain Monte Carlo Methode für die Photonenerzeugung[6] generiert. Die Donor und Akzeptor Photonenzahl definiert die Bursteffizienz, aus der dann in Analogie zum Experiment Bursteffizienzhistogramme gebildet werden, und mit den experimentellen Histogrammen verglichen werden können. Unsere Simulationen zeigen, daß es zwei Gründe für die experimentell beobachtete Heterogenität gibt. Zum einen hat Poly-Prolin eine nicht zu vernachlässigende Isomerisierungswahrscheinlichkeit. Daher sind Teile des Ensembles cis-Isomere mit einem geringeren End-zu-End Abstand. Ausserdem wechselwirken die Farbstoffe mit dem Poly-Proline Rückgrat was zu offenen und geschlossenen Zuständen mit unterschiedlicher Dynamik und Lebensdauer führt [3].
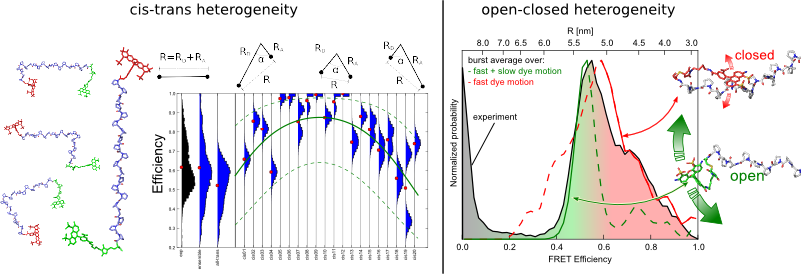
Konformationsheterogenitaet
Links: Effizienzhistogramme aller all-trans und cis Isomere. Cis-Bindungen in zentraler Position verringern den Abstand und führen daher zu einer erhöhten FRET Effizienz. Rechts: Effizienzhistogramme der schnellen und langsamen Farbstoffbewegung, welche ihren Ursprung in offenen und geschlossenen Konformationen haben, und deren Vergleich mit dem Experiment.
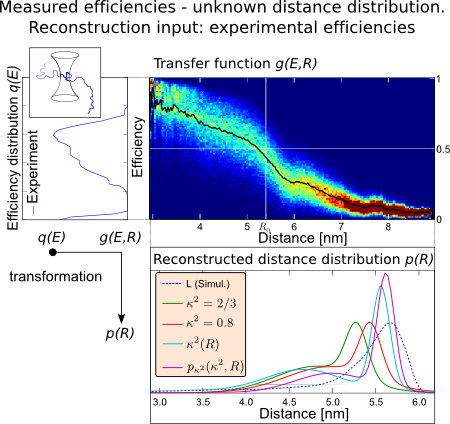
Rekonstruktion
Abstandsbestimmung
Als nächstes wurde eine Methode zur Rekonstruktion der Abstände auf Grundlage von Transferfunktionen \(g(E,R)\) \[q(E) = \int\limits_0^{R_{max}} g(E,R) p(R) dR\quad,\] mit \(q(E)\) als Effizienz- und \(p(R)\) als Abstandsverteilung entwickelt. Der Transferfunktionsformalismus beschreibt eine Faltung der Abstandsverteilung mit einen beliebigen Faltungskernel. Dieser ist im einfachsten Fall die Förster Gleichung (1). Ausgehend von diesem wurde der Faltungskernel durch systematischer Reduktion der Annahmen in Gl. (1) angepasst. Dabei wurden die Annahmen durch die Orientierungsstatistik und -dynamik der MD Simulationen ersetzt [3]. Die rechte Seite zeight ein Rekonstruktionsbeispiel. Dabei wird die aus dem Experiment bestimmte Effizienzverteilung durch die Transfermatrix (Regenbogenfarben) in eine Abstandsverteilung überführt. Die Abstandsverteilungen sind unten im Bild für verschiedene Näherungsstufen gezeigt. Daraus geht hervor, daß durch jede Reduktion der Annahmen eine systematische Verbesserung der Genauigkeit der rekonstruierten Abstände erzielt werden kann.
Literaturverzeichnis:
- Förster, T. Zwischenmolekulare Energiewanderung und Fluoreszens Ann. Phys., 1948, 2, 55-75
- VanBeek, D. B.; Zwier, M. C.; Shorb, J. M. & Krueger, B. P. Fretting about FRET: correlation between kappa and R. Biophys J, 2007, 92, 4168-4178
- Hoefling, M.; Lima, N.; Hänni, D.; Schuler, B.; Seidel, C. A. M. & Grubmüller, H. Structural Heterogeneity and Quantitative FRET Efficiency Distributions of Polyprolines through a Hybrid Atomistic Simulation and Monte Carlo Approach PLoS ONE, 2011, 6, e19791
- Stryer, L. & Haugland, R. P. Energy transfer: a spectroscopic ruler. Proc Natl Acad Sci U S A, 1967, 58, 719-726
- Schuler, B.; Lipman, E. A.; Steinbach, P. J.; Kumke, M. & Eaton, W. A. Polyproline and the “spectroscopic ruler” revisited with single-molecule fluorescence. Proc Natl Acad Sci U S A, 2005, 102, 2754-2759
- Pool, R.; Heringa, J.; Hoefling, M.; Schulz, R.; Smith, J. C. & Feenstra, K. A. Enabling grand-canonical Monte Carlo: Extending the flexibility of GROMACS through the GromPy python interface module. J Comput Chem, 2012